
A partir du nom de la protéine trouver la séquence d'acides aminés, visualiser la structure 3-D, trouver des séquences similaires et construire un arbre phylogénétique.
N.B: pour accéder au site du cours : tapez http://doiop.com/bist
Rappels : Ce qu'est une protéine :
Lire le document "Steak ou haricots secs?" sur prolune
Les 20 acides aminés et leur structure
Exploiter le document sur expasy (pdf)
Trouver la séquence d'une protéine à partir de son nom dans la banque de donnees UniProt (SwissProt) Des exemples de protéines amusantes.pdf
Convertisseur code 1 lettre -> code 3 lettres des acides aminés : One -to-Three | Three to One
Tableau des Abbrév acides aminés à une lettre/ 3lettres
Autres activités possibles :
|
Pour une découverte plus authentique / pour aller plus loin : Trouver l'insuline dans la banque de données UniProt (SwissProt) : Solution On voit que c'est le précurseur de l'insuline, qu'il contient 2 chaînes, que son nom abrégé est INS En principe Toooooouuuuuut en bas on trouve la séquence :
10 20 30 40 50 60
MALWMRLLPL LALLALWGPD PAAAFVNQHL CGSHLVEALY LVCGERGFFY TPKTRREAED
70 80 90 100 110
LQVGQVELGG GPGAGSLQPL ALEGSLQKRG IVEQCCTSIC SLYQLENYCN
|
Lancer par le web le programme 3-D Structure browser pour écoles (merci SwissProt)
De la séquence à la structure 3-d : Conseils et mode d'emploi (pdf)
Trouver la structure 3-d de l'insuline : cliquer sur l'icône I ![]() à côté de "insuline "
à côté de "insuline "
|
Pour afficher d'autres modèles de visualisation : Cliquer à droite (ctrl-clic sur Mac) et choisir Sélectionner -> tout , puis Rendu choisir le modèle dont vous avez l'habitude (p.ex. ->Combinaison > CPK remplissage.) |
L'orienter de manière à bien voir les ponts disulfures qui relient les 2 chaînes: exemple ils sont en jaune ici : 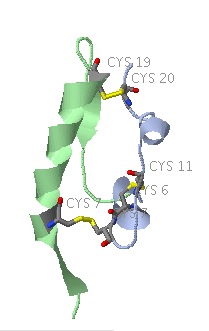 Copier l'image (Capture sur pc , pomme-shift-4 sur Mac) l'intégrer dans le document à rendre.
Copier l'image (Capture sur pc , pomme-shift-4 sur Mac) l'intégrer dans le document à rendre.
Afficher les protéines suivantes
et chaque fois :
| Pour aller plus loin ( cf scénario 4) |
Principe
Document de Référence : conseils et mode d'emploi (pdf)
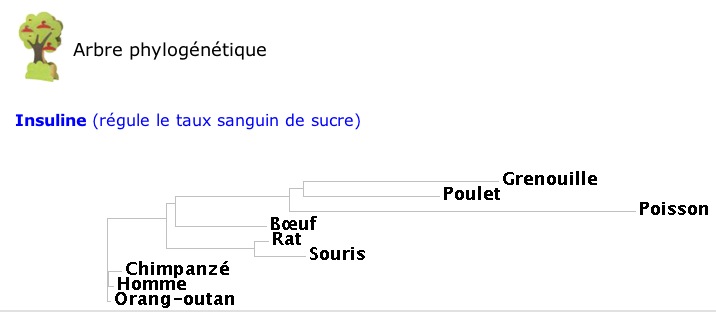
A) Trouver pour la proétine qui vous intéresse, des séquences équivalentes chez de nombreuses autres espècesPour cela, il faut utiliser un programme bioinformatique (appelé ‘Blast’) qui va ‘pêcher’ dans une base de données toutes les protéines qui ressemblent à la protéine de départ. La base de données que nous utilisons est appelée Swiss-Prot. Elle contient des informations sur ~230’000 protéines provenant de ~10’000 espèces différentes.
On obtient une liste de séquences alignées.
Noter quels groupes d'êtres vivants sont représentés et lesquels sont absents.
Observer les zones qui sont différentes chez la plupart des organismes et celles qui varient peu. On parle de zones conservées pour les zones de la protéine qui ne varient pas d'une espèce à l'autre.
Repérez ces zones conservées et essayez d'imaginer pourquoi certaines zones changent plus que d'autres.
|
Pour aller plus loin Scénario 5 |
Retour à BIST | SwissProt| M.C. Blatter | Projets Home de F. Lo