
A partir du nom de la protéine (Erythropoiétine EPO) localiser le gène sur les chromosomes humains, trouver la séquence d'acides aminés et éventuellement la séquence en acides nucléiques.
Pour une version simplifiée voir BIST-ABC
La démarche inverse, du gène dans les chromosomes à la séquence du gène : scénario 1b iciCet exemple a été réalisé avec l'EPO , mais on peut le réaliser avec l'insuline ou avec d'autres protéines : Liste d'exemples de protéines
Swiss-Prot Banque de données de séquences de protéines annotées manuellement . Swiss-Prot et TrEMBL (annotation automatique) réunis s'appellent UniProt et donnent accès à toutes les séquences de protéines connues (~18Millions).
Accéder à UniProt (=Swiss-Prot),
Convertisseur code 1 lettre -> code 3 lettres des acides aminés : One -to-Three | Three to One
Tableau des Abbrév acides aminés à une lettre/ 3lettres
Ce browser indique dans une page interactive ![]() de nombreuses informations dont :
de nombreuses informations dont :
la séquence présentée en bases : chr7:100,318,423-100,321,323 2,901 bp
un schéma du chromosomes correspondant ( 7) avec la position repérée en rouge ( bras long vers le milieu)

les introns représentés avec une barre épaisse et les exons avec le sens de transcription indiqué par des >>>> ou <<<<

On peut visualiser les variantes et les snp en ajustant les paramètres Touuuut en bas : common SNPs(138) : full et Genome variants : Full

On peut ainsi voir l'alignement avec d'autres organismes et les variantes connues.
Le degré de conservation de la protéine est un reflet de la pression de sélection.

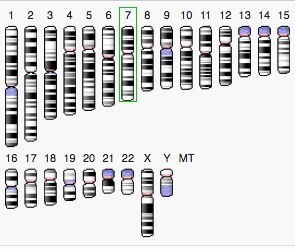 L'idéogramme à gauche montre un schéma des chromosomes avec le chromosome 7 encadré codant pour l'EPO :
L'idéogramme à gauche montre un schéma des chromosomes avec le chromosome 7 encadré codant pour l'EPO :
cf ci-contre : cliquer pour agrandir .
Partie de droite : Le schéma en haut représente le chromosome 7 avec en bleu pale la position de la zone agrandie en-dessous.
Les bandes en-dessous schématisent une section du chromosome avec le gène EPO au milieu la séquence, la structure du gène (introns-exons) et d'autres paramètres comme les SNP
Sélectionner dans la bande (avec les position en nucleotides ou K) pour effectuer un zoom 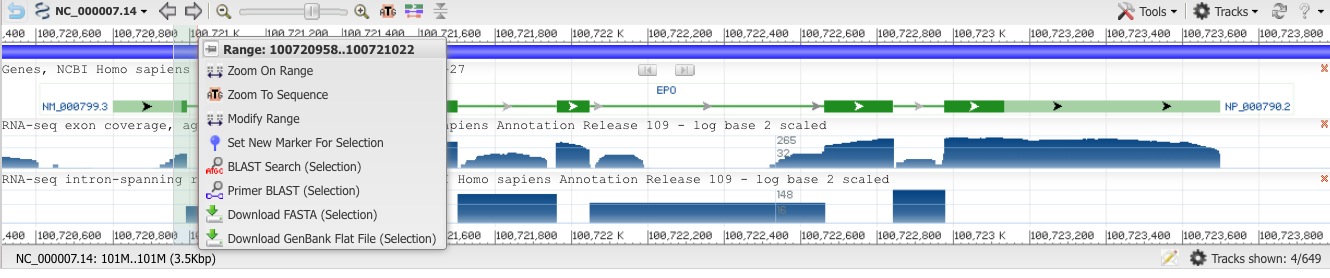 : Selectionner l'icone zoom to selection pour Zoomer "out" jusqu'à faire apparaitre la limite entre un exon et un intron pour noter qu'ils ne sont pas intrinsèquement différents. (solution)
: Selectionner l'icone zoom to selection pour Zoomer "out" jusqu'à faire apparaitre la limite entre un exon et un intron pour noter qu'ils ne sont pas intrinsèquement différents. (solution)
On peut aussi zoomer en arrière (dans le schéma du chromosome et visualiser l'ADN non codant entre les gènes et mettre en évidence la faible place des gènes. (solution)
On peut aussi faire cette procédure avec d'autres protéines connues.
M. C. Blatter a rassemblé une Liste d'exemples de protéines avec :
Retour à BIST | Swiss-Prot| M.C. Blatter | Projets Home de F. Lo