
| Version simplifiée : 3-D Structure browser spécial écoles Documentation (merci SwissProt). |
être capable de visualiser la structure 3-D d’une protéine (si elle est connue)
Y repérer des zones pertinentes à l'activité ou aux mutations
Il y a différentes façons de visualiser la structure 3D d'une protéine. Dans nos écoles, installer un logiciel est souvent difficile. Nous choisirons une méthode qui semble fonctionner dans la plupart des cas.
Ce qu'il faut savoir: -
1. Il y a très peu de protéines dont la structure 3D est connue (déterminée expérimentalement par cristallographie ou RMN) (environ 6000 sur 160'000 protéines répertoriées dans Swiss-Prot)
2. Il y a une seule banque de données concernant les structures 3D des protéines, des sucres et de l'ADN: PDB pour protein data bank. Cette banque contient pour chaque molécule cristallisée, les coordonnées spatiales de chacun des atomes. Important: elle ne contient pas de modèle (ou image) 3D.
3-D :. Pour avoir une image (un modèle) 3D d'une molécule on doit utiliser des logiciels (3d-Viewers), plus ou moins compliqués qui utilisent les données de PDB pour construire des modèle: Chime, Rasmol...etc. On peut aussi utiliser des outils disponibles directement dans Swiss-Prot par exemple (qui ne nécessitent pas d'installer un programme sur sa machine).
Depuis la home page du serveur UniProt: et taper INS_HUMAN, puis "Search"
Vous arrivez (solution) dans la fiche UniProt décrivant l'insuline humaine.
Trouver dans la page, rubrique Cross-references, les liens vers les structures 3D = PDB :
il y a en plusieurs, car l'insuline a été cristallisée de nombreuses fois.
Choisir une des entrées PDB (au hasard) puis cliquer sur RCSB : Solution exple .
On voit à droite dans un encart la molécule et on peut y cliquer le mode de visualisation
Dans Display options, cliquer JMol (Solution )
On peut "tourner la molécule virtuellement, cliquer à droit sur la molécule pour les options de visualisation
Les memes molécules peuvent être visualisées par un autre programme de visualisation : QuickPDB : on choisit la protéine en recopiant son numéro 1BEN ou 1A7F poour l'insuline dans le champ (Solution)
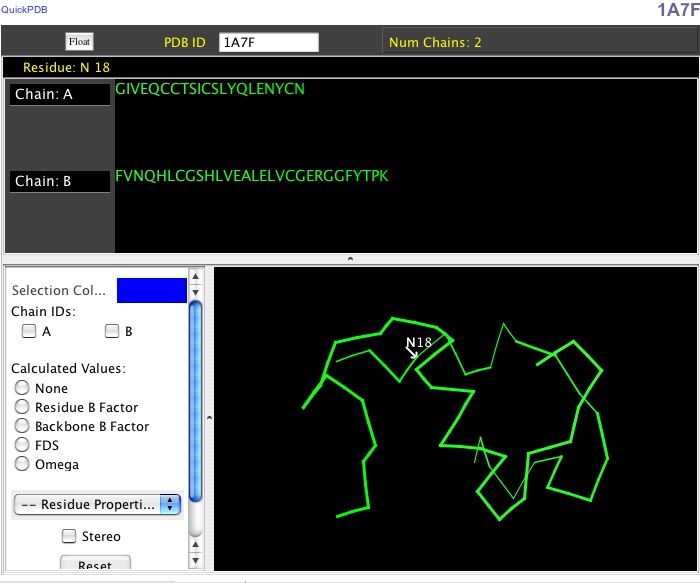
On y voit la séquence en meme temps que la structure et quand on survole avec le curseur de la souris une partie de la séquence dans le panneau du haut , une flèche indique la zone correspondante de la structure et vice versa : si on clique sur la structure, l'a.a. correspondant s'"allume".
PDB pour protein data bank. Cette banque contient pour chaque molécule cristallisée, les coordonnées spatiales de chacun des atomes. Elle ne contient pas de modèle (ou image) 3D
EBI (Un serveur qui met à disposition un viewer très complet Aztex Viewer) (Solution)
Dans un menu à gauche de la page, cliquer sur "Visualisation"
On vous propose une liste de viewers (programmes de visualisation). La plupart doivent être installés sur votre machine avant de pouvoir être utilisés. En général il est plus facile d'en choisir un qui n'a pas besoin d'être installé.
Java est un langage de programmation qui produit des logiciels qui tournent sur toutes les machines courantes (Mac, PC, Linux, *unix). AstexViewer nécessite que Java soit activé sur votre ordinateur.
Trouver le lien "View the PDB entry using ...." qui lance une applet java ( petit programme).
( il fait bien cliquer le mot view ...sinon on a droit à leur pub'
Le lancement de l'applet peut prendre plusieurs dizaines de seconde selon la liaison.
 Vous pouvez
Vous pouvez
Le plus simple pour trouver d'autres exemples de structures 3D est d'utiliser le portail 'Molecule of the month' de PDB
Pour chaque protein il y a un petit texte (en anglais) qui explique la structure.
Connaissant le numéro d'accession dans PDB (ex 1GFL) on peut aller chercher l'entrée Swiss-Prot correspondante avec 1GFL sur UniProt) afin de suivre le lien vers EBI pour avoir accès à Astex viewer.
Voici quelques autres protéines
Comme Swiss-Prot ne traite que les protéines, les ARN de transfert (phenylalanine 4tna ou Aspartate 2tra) ne sont visibles que dans PDB avec leurs viewers (essayer Jmol) . (les anticorps n'y sont pas traités a cause de leur nombre énorme) ..
Depuis la home page du serveur UniProt:
Utiliser l'outil de recherche "Swiss-Prot/TrEMBL protein" y taper HBB_HUMAN, puis "GO" Solution
Tout en bas de la page, de la fiche Swiss-Prot, sous le bandeau bleu FEATURES lil y a une liste de VARIANT, des principales mutations trouvées dans HBB.
Trouver, par exemple, le Variant en position 6, responsable de la "sickle cell anemia"
VARIANT 6 6 1 E -> V (in S; sickle cell anemia; dbSNP:334) [NCBI/Ensembl]. VAR_002863 [3D]
Cliquer sur VAR_002863 Vous arrivez sur une page décrivant en détail la mutation. (Solution)
Advanced : On y apprend que ce changement en position 5 a comme effet Change from medium size and acidic (E) to medium size and hydrophobic (V)
Trouver vers le mlieu de cette page "Model visualization"
Cliquer sur "Astex viewer", ![]() pour une visualisation 3D de cette molécule (Solution)
pour une visualisation 3D de cette molécule (Solution)
Vous pouvez déplacer dans l'espace le modèle, et voir tantôt la séquence "wild" tantôt la séquence "mutée" en cliquant sur les acides aminés au-dessous où les 2 séquences sont présentées.

Cliquer sur le 6 ( Y ou E) montre bien la différence entre le variant et le Type sauvage
N.B: le mode de visualisation ne semble pas pouvoir être changé lors de comparaisons de 2 séquence.
Ce type de démarche peut-être faite pour tous les variants décrits dans Swiss-Prot et pour lesquels un modèle a été calculé. On les repère dans la ligne des Variant au tag [3D]
Par exemple
VARIANT 6 6 1 E -> V (in S; sickle cell anemia; dbSNP:334) [NCBI/Ensembl]. VAR_002863 [3D]
Retour à BIST | Swiss-Prot| M.C. Blatter | Projets Home de F. Lo -->