
P. ex. une protéine présente chez de très nombreuses espèces pour construire l'arbre de la vie (p. ex. un facteur d'élongation EF1A). Une sélection de séquences pour cette protéine chez des espèces bien diversifiées est disponible ici (format FASTA)
Pour plus info sur ces séquences aller voir les entrées sur le site UniProt. Il s’agit donc de séquence de facteur d’élongation (appelé EFTU ou EF1A). On voit assez facilement appraitre la séparation eucaroytes, archae et bactérie. L'arbre produit par Phyolgeny.fr avec ces données.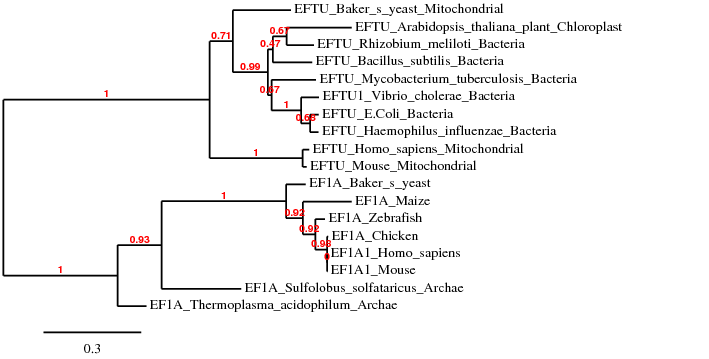
Phylogramme pour EFTU / EF1A, en rouge les "Branch support value" c'est à dire l'indice de confiance qu'on peut accorder à cette bifurcation. (version.pdf)
Remarques : certaines branches sont très fiables comme la première bifurcation qui correspond à un évènement de duplication de gène avec passage nucléaire - plastide (1) alors que d'autres sont moins certains comme la bifurcation entre levure et maïs.
On observe que certaines branches sont différentes, ce sont justement celles qui ont un "Branch support value" restreint ; (moins de 0.9)
+---EFTU_BACSU
+--7
| | +--EFTU1_VIBCH
| +--8
| | +EFTU_HAEIN
| +--9
| +EFTU_ECOLI
|
+--5 +----EFTU_MYCTU
| | |
| | | +---EF1A_MAIZE
| | | +---------12
| | | | | +---EF1A_YEAST
| | | | +-13
| | | | | +EF1A_DANRE
| +--4 | +-14
| | +-11 | +EF1A_CHICK
| | | | +-15
| | | | | +EF1A1_MOUSE
| | | | +-16
| | +-----------------------------10 | +EF1A1_HUMAN
| | | | |
| | | | +--------EF1A_SULSO
| +--3 |
| | +----EF1A_THEAC
| |
| | +-------EFTU_YEAST
| +--2
| | +EFTU_MOUSE
| +-----------1
| +EFTU_HUMAN
|
6--------EFTU_ARATH
|
+---EFTU_RHIME
N.B: Les séquences EFTU_ARATH (arabidopsis) EFTU_YEAST, EFTU_MOUSE et EFTU_HUMAN sont dérivées des génomes chlorophastiques et mitochondriaux respectivement. Ces séquences sont donc plus proches des séquences de bactéries que des eucaryotes dans l’arbre
Pour le contexte : cet exemple complète la bio-review sur les phéromones de janvier 08 :
Partir d'un des gènes de récepteur humain aux phéromones VN1R5
Le BLAST produit une page avec les séquences et un schéma coloré du degré de similitude et de la colinéarité.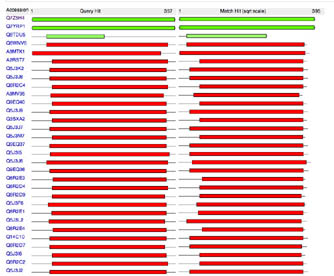

Objectif: Explorer les fonctions biologiques caractéristiques qui ne seraient présentes que dans certains règnes et vérifier ces hypothèses à l'aide du Blast : explorer quelle protéine employer pour établir le cladogramme. Quelle protéine et universelle ou au contraire spécifique d'un règne.
Attention: ce n'est pas parce qu'une séquence n'est pas présente pour une espèce donnée qu'elle n'existe pas ! (mais ce type d'approche permet quand même de se faire une bonne idée de la répartition 'taxonomique' de certaines fonctions biologiques..)
Pour cela faire un Blast avec une protéine à explorer (liste d'exemples) sur ExPASy Pas encore sur UniProt car Blast@expasy a des options qui permettent entre autre d'avoir accès à une vue globale de la distribution taxonomique des résultats
Il y a 2 options:
1 - Plus facile à lire : Choisir dès le départ un groupe taxonomique pour faire le Blast (Taxonomic groups: select a database subsection) (utile par exemple pour vérifier que la protéine n'existe pas dans un groupe particulier, exemple chez les procaryotes)
2 - Plus exhaustif : Faire un Blast contre toute la banque de donnée (pour faire plus simple choisir de toute façon l'option 'Search only Swiss-Prot (curated sequences)' et regarder la 'Taxomony view' (la distribution parmi les différentes espèces).
Retour à BIST | Swiss-Prot| M.C. Blatter | Projets Home de F. Lo