


Glossaire
- A) Moteurs de recherche
- B) 10 principales banques de données
- C) Numéro d'accession
- D) Noms abrégés des espèces
- E) Termes
A) Moteurs de recherches utilisés dans ce cours
Exemple: si on cherche des informations sur l'insuline, on trouvera certainement des données dans toutes les banques citées. Le moteur de recherche sélectionnera les entrées en nous donnant une liste cliquable de no d'accession.
Selon le type de données qu'on cherche on accède aux données par différents portails :
- Entrez Genome : Mapviewer toutes les espèces, pour l'homme:

- Entrez Protein

- Entrez Nucleotide

- Entrez Gene

- Entrez PubMed

- Gene and Diseases

A la base un ouvrage de référence sur de nombreuses maladies génétiques devenu un portail spécifique
B) les principales banques de données
Pour chacune des banques, vous trouverez l'adresse web, ainsi qu'une brève description de son contenu.
PubMed http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?db=PubMed
Banque de données bibliographiques: tous les articles publiés dans le domaine de la biologie et médecine sont répertoriés depuis 1960 environ.
OMIM http://www.ncbi.nlm.nih.gov/Omim/searchomim.html 
Banque de données génomiques: donne des informations sur les gènes humains, leur localisation sur le génome et avec quelles maladies ils sont associés.
EntrezGene http://www.ncbi.nih.gov/entrez/query.fcgi?db=gene
Banque de données qui donne des informations sur les gènes humains et d'autres espèces, leur localisation sur le génome et des informations sur leurs fonctions.
RefSeq http://www.ncbi.nlm.nih.gov/RefSeq/
Banque de données de séquences en acides nucléiques et protéiques. RefSeq choisit pour chaque gène, une séquence de référence pour l'ADN (séquence génomique) une séquence de référence du mRNA, une séquence de référence pour la protéine.
Swiss-Prot http://www.uniprot.org/
Banque de données de séquences de protéines annotées manuellement et regroupant un maximum d'information biologiques et de liens vers les autres banques de données. TrEMBL, la grande soeur de Swiss-Prot contient toutes les séquences de protéines qui ne sont pas encore dans Swiss-Prot, parce que pas encore annotées manuellement. Swiss-Prot et TrEMBL réunis s'appellent UniProt et donnent accès à toutes les séquences de protéines connues.
HMGD (Human gene mutation database) http://archive.uwcm.ac.uk/uwcm/mg/hgmd0.html 
Banque de données donnant accès aux mutations connues d'un gène, responsables d'une maladie
Newt http://www.ebi.ac.uk/newt/index.html Banque de données donnant des informations sur la classification des organismes
C) Numéro d'accession (AC)
Accès unique aux infos concernant un gène ou une séquence.
Chaque banque de données a des numéros d'accession différents. A priori cela ressemble à une jungle, mais une fois que l'on est habitué, cela permet de savoir dans quelle banque on se trouve.
- RefSeq:
NM_ pour les mRNAs,
NT_ pour les séquences génomiques, (aussi NC_XXXXX, NG_XXXXX, NW_XXXXXXX),
NP_ pour les protéines.
Tout ce qui commence par X (XM_XXXXX, XP_XXXXX) sont des entrées dérivées de prédiction (pas de preuve expérimentale).
- EntrezGene:
LocusID (par exemple 3630) : .
- Swiss-Prot/TrEMBL:
ONNNNN, PNNNNN, QNNNNN ; par exemple : P12345
Codes nucléotides inhabituels
En plus des 4 bases A T C G
- N : N'importe quelle base
- R : A ou G (purine)
- Y : C ou T (Pyrimidine)
- - : --- aucune (gap = lacune)
nCodes acides aminés inhabituels
En plus des 20 acides aminés (code à une lettre ) on trouve (1-lettre, puis 3-lettre)
- B Gln ou Glu
- Z Asn ou Asp
- X Xaa n'importe quel résidu
- - --- aucun résidu correspondant (gap = lacune)
Petits carrés de couleur
 G Entrez Gene
G Entrez Gene
 U UniGene
U UniGene
 E Entrez GEO Gene Expression Omnibus
E Entrez GEO Gene Expression Omnibus
 O OMIM
O OMIM
Anciens ? obsoletes ?
 N Nomenclature
N Nomenclature
 R RefSeq
R RefSeq
 G GenBank
G GenBank
 P Protein
P Protein
Noms des espèces en abrégé (Code à 2 et à 5 lettres)
Dans certaiens banques le nom des organismes est abrégés en 2 lettes (Ce, Mm, Hs, etc) ou 5 lettres ("BRARE")
Code à 2 lettres
Le code 2 lettres des organismes est composé des premières lettres des noms complets
- Hs: Homo sapiens (human)-> HUMAN
- Mm: Mus musculus (mouse)-> MOUSE
- Dm: Drosophila melanogaster (fruit fly)-> DROME
- Ce: Caenorhabditis elegans (nematode)-> CAEEL
- Rn: Rattus norvegicus (rat)-> RAT Sc: Saccharomyces cerevisiae (Yeast) -> YEAST
- Br: Brachydanio rerio (Zebrafish) (Danio rerio)- BRARE
Code 5 lettres
Le code 5 lettres code est composé des 3 première lettres du premier mot et des 2 premières lettres du second (il est spécifique à Swiss-Prot),
Exemple: PSEPU = Pseudomonas putida and NAJNI = Naja nivea.
Exception: les 16 espèces les plus utilisées dans la recherche
- BOVIN for Bovine,
- CHICK for Chicken
- ECOLI for Escherichia coli
- HORSE for Horse,
- HUMAN for Human,
- MAIZE for Maize (Zea mays),
- MOUSE for Mouse,
- PEA for Garden pea (Pisum sativum),
- PIG for Pig, RABIT for Rabbit,
- RAT for Rat,
- SHEEP for Sheep,
- SOYBN for Soybean (Glycine max),
- TOBAC for Common tobacco (Nicotina tabacum),
- WHEAT for Wheat (Triticum aestivum), and
- YEAST for Baker's yeast (Saccharomyces cerevisiae).
Les noms des organismes abrégés (PANTR, tels que p. ex. ici )
se trouvent dans la liste des espèces citées dans Swiss-Prot http://www.expasy.org/cgi-bin/speclist
Glossaire de termes
-
- Bandes chromosomiques
- Encore à finir mais en relation avec la coloration de l'eu / hétéro chromatine
- Base / banque de données
- Sorte d'Encyclopédie informatique regroupant un grand nombre de données notamment de séquences mais aussi de bibliographie etc...
- Bioinformatique
- Utilisation d'outils informatiques pour gérer et analyser des données biologiques.
- BLAST
- outil informatique permettant de faire des recherche de similarité à partir d'une séquence (protéine ou nucléique) sur toutes les séquences (ou un sous-ensemble de séquences) existantes.
- Nucleotide-nucleotide BLAST (blastn)
- Protein-protein BLAST (blastp)
- Translated query vs. protein database (blastx)
- Protein query vs. translated database (tblastn)
- Translated query vs. translated database (tblastx)
- Branch support value
- Probabilité que la bifurcation soit existe sous cette forme.
-
- Browser (Navigateur, butineur)
- Un programme informatique permettant de visualiser des informations provenant du web (Firefox, Firefox, Internet Explorer, Mozilla, Safari, Netscape,...).
-
- cDNA
- Par convention, le cDNA est la copie conforme du l'ARN m (T remplaçant U); c'est une notion purement "in silico": un cDNA n'existe pas dans la cellule à proprement parler.
Ce n'est donc pas le brin lu ou brin matrice notamment à cause des introns en moins, poly A en plus...
-
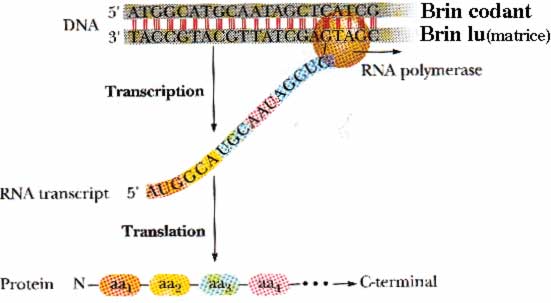
- -D'autres (source) le définissent comme ADN simple brin, qui est une copie d'un ARN obtenue par une transcription inverse.
- Ce n'est alors pas le brin lu ou brin matrice mais par convention le brin "sens" ou brin codant
- La séquence d'un gène (DNA génomique) correspondant à la séquence du mRNA (T remplaçant U et les introns en moins, poly A en plus....)
Le DNA génomique est donc également une copie "conforme" (du point du vue du sens de la séquence; i.e. il ne s'agit pas de la séquence inverse) du cDNA donc de l'ARNm.....
-
- Celera
- Le génome humain a été séquencé par 2 grands consortium : " public " et " privé /Celera ".
Le projet public a pris comme " donneurs " une quinzaine de personnes dont seules quelques unes (absolument anonymes) ont finalement été ‘séquencées’. Le projet privé a séquencé le génome de M. Craig Venter, directeur de Celera (pour l’anecdote, il a aussi fait séquencer le génome de son chien, un caniche).
Les 2 consortiums ont publié respectivement dans Nature (projet public) et Science (projet privé) en 2000 les résultats de leur premier " brouillon " du génome complet. Ils viennent tous les 2 de publier à nouveau les résultats d’un nouveau nettoyage et assemblage des séquences. Les séquences dérivées du consortium privé étaient restées longtemps non-publiques. Elles sont actuellement intégrées dans les données publiques.
- CoDing Sequence (CDS)
- Région d'un mRNA codant pour une protéine, comprise entre le codon codant pour la première méthioinine et le codon STOP.
- Epissage (splicing, alternative splicing)
- (Eucaryotes) processus (impliqué dans la mutaration des ARNm) au cours duquel un ARN est modifié (élimimation des introns et de certains exons). Si un même ARN peut subir différentes modifications, on parle d'épissage alternatif.
- Fasta
- (format fasta) : Format (convention d'écriture) d'une entrée dérivée d'une banque de séquences qui contient un minimum d'information et qui est compatible avec la plupart des outils informatiques. Une entrée en format Fasta commence par > puis le nom du gène ainsi que de l'organisme duquel il est dérivé, suivi de la séquence (ADN ou protéine) en « raw data » : les lettres du code à une lettre en majuscule .
- Exemple :
>HSGLTH1Human theta 1-globin gene
CCACTGCACTCACCGCACCCGGCCAATTTTTGTGTTTTTAGTAGAGACTAAATACCATATAGTGAACACCTAAGA
CGGGGGGCCTTGGATCCAGGGCGATTCAGAGGGCCCCGG....
- Hyperlinks (cross-links, cross-références)
- liens cliquables qui permettent de passer d'une banque de données à une autres.
- Gène
- morceau d'ADN qui code soit pour une protéine (via un ARNm) soit pour un ARN fonctionnel (ARN ribosomaux, ARN de transfert, micro ARN etc...).
- Région d'ADN 'codante' pour des protéines ou des ARN fonctionnels: les 2 sont souvent (mais pas toujours) différenciés dans les statistiques des génomes.
- Locus
- (loci au pluriel) : position d'un gène sur un chromosome.
- Modifications post-traductionelles des protéines (PTMs)
- Une fois synthétisée, une protéine peut subir de nombreuses modifications : addition de sucres (glycosylation), addition de phosphates (phosphorylation), addition de lipides, clivage d'un signal ou d'un propeptide, formation de ponts dissulfures?etc?Il existe environ 200 différentes PTMs.
- Moteur de recherche
- Outil informatique permettant de trouver des informations dans plusieurs banques de données à partir d'un nom de gène ou d'un nom de maladie (exemples : Entrez, Mapviewer, Gene and diseases).
- Numéro d'accession
- Numéro unique ou code qui identifie une entrée dans une banque de données. Ces numéros sont spécifiques pour chaque banque de données et restent stables.
- Homologue
- Protéines ou gènes qui sont similaires et qui ont une origine commune.
- Des protéines homologues découlent de la divergence de 2 gènes dérivés d'un ancêtre commun. Il ne s'agit pas d'un synonyme de « similaire » : 2 protéines similaires ne sont pas forcément dérivées d'un ancêtre commun.
- Une similitude de plus de 25% au niveau des acides aminés (25 acides aminés identiques sur 100) et de plus de 70% au niveau des nucléotides est considée comme une "homologie" ( Claverie & Notredame p 229)
- Paralogue
- Protéine homologues qui ont des fonctions différentes mais liées, au sein d'un même organisme.
- Orthologue
- Protéines homologues qui ont des fonctions différentes mais liées, au sein d'organismes différents.
- Polymerase chain reaction (PCR)
- technique de biologie moléculaire qui permet de photocopier une séquence spécifique d'ADN.
- Portail
- Site web et outil informatique associé à une banque de données permettant de rechercher et de visualiser les informations qui s'y trouvent.
- Protéome
- Ensemble des protéines d'un organisme vivant.
- Signal
- Séquence d'une protéine (en N-terminal) qui permet de « diriger » la protéine nouvellement synthétisée dans la voie secrétoire de la cellule. Le signal est clivé dans la forme mature de la protéine.
- Serveur
- un puissant ordinateur relié au réseau, localisé dans un institut spécialisé, donnant accès à des banques de données, des outils permettant d'utiliser ces banques de données ou des outils plus spécifiques. Dans le domaine de la biologie il y a beaucoup de serveurs disponibles, mais 2 d'entre eux sont beaucoup utilisés:
Chacun de ces serveurs a ses spécialités, les banques mises à disposition et les outils ne sont pas forcéments les mêmes, ainsi que les mises à jour.
- ExPASy est spécialisé dans le domaine des protéines.
- NCBI donne accès aux séquences en acides nucléiques et protéiques.
UniGene
- UniGene: regroupe dans une même entrée toutes les séquences (ARN) dérivées d'un même gène et ajoute des informations comme la similitude avec d'autres protéines ou la localisation sur le génome.
Autres glossaires


![]()
![]()
![]()
![]()


